植物分子生理学・年報(2024年度)
研究内容の概略
本研究室では、植物が示す驚異的な環境適応能力の分子基盤として、環境刺激に応答したゲノム規模の遺伝子発現制御、およびその結果もたらされるプロテオームの多様化やオルガネラの機能分化、また細胞・組織・器官間で行われる長距離シグナル伝達などの過程に着目し、それらの現象を遺伝子、タンパク質および細胞レベルで研究しています。その際、ゲノム科学、分子遺伝学、分子生物学、生化学、細胞生物学、植物生理学などの手法を複合的に駆使し、多面的なアプローチによって研究を展開しています。
1. 転写開始点選択の仕組みと遺伝子機能の多面性
生物の複雑さはプロテオームの多様さに依存しますが、ある1つの生物種が持つ遺伝子の数には限りがあります。そこで、より高度な生命活動を営むためには、機能の異なる複数のタンパク質を1つの遺伝子から生み出す仕組みが必要となります。
転写開始点選択とは、1つの遺伝子内に存在する複数の転写開始点から、長さの異なるmRNA分子が転写される現象のことであり、選択的スプライシングと並んで、プロテオームの拡大に貢献しうる機構として知られています。しかしながらこれまで、プロテオームに対するインパクトは小さいと考えられていたため、その重要性は軽んじられてきました。
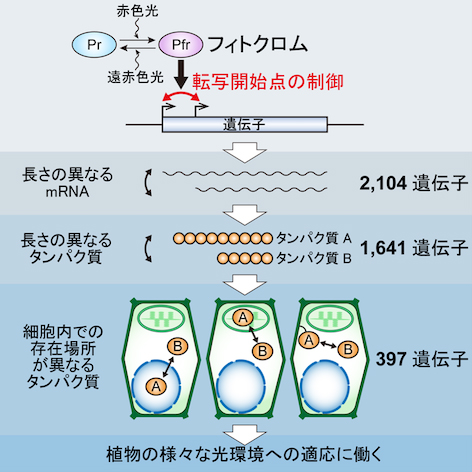
私たちは最近、植物の主要な光受容体であるフィトクロムが、シロイヌナズナにおいて2,000を超える遺伝子に直接働きかけそれらの転写開始点を変化させること、これに伴い約400のタンパク質の細胞内局在が光によって変化すること、そしてそれらタンパク質の細胞内局在変化が植物の様々な光環境への適応に寄与することを発見しました(Ushijima et al., Cell 2017)(図1)。
これらの発見は、転写開始点選択という現象が、転写・スプライシング・翻訳と並び、真核生物のセントラルドグマにおける新たな普遍的一過程として、プロテオームの機能的な多様化に少なからず寄与することを強く示すものであります。そして同規模の転写開始点変化は、フィトクロムシグナルに限らず、ありとあらゆるシグナルにより、真核生物において共通の分子機構で引き起こされるものである可能性が高いと考えられます。
そこで私たちは、モデル植物であるシロイヌナズナを用いて、フィトクロムによる転写開始点制御をモデルケースとしてその分子機構を解明することで、真核生物に普遍的な新規遺伝子発現制御機構を明らかにし、セントラルドグマに新たな一過程を付け加えることを目指します。その結果として近い将来、生物の教科書の書きかえが行われるものと期待されます。
また、様々な環境刺激に応じて転写開始点が変化することで、同じ1つの遺伝子から、これまで知られていた機能とは全く異なる機能を持ったタンパク質が生じるケースが、次々と明らかになってきました。そこで今後私たちは、さらに多くの遺伝子について、転写開始点の切り替えによって発揮される遺伝子機能の多面性を明らかにし、多くの遺伝子が持つ「裏の顔」を暴くことで、プロテオームの未開領域の開拓を進めます。
2. 植物の芽生えにおける環境応答の解析
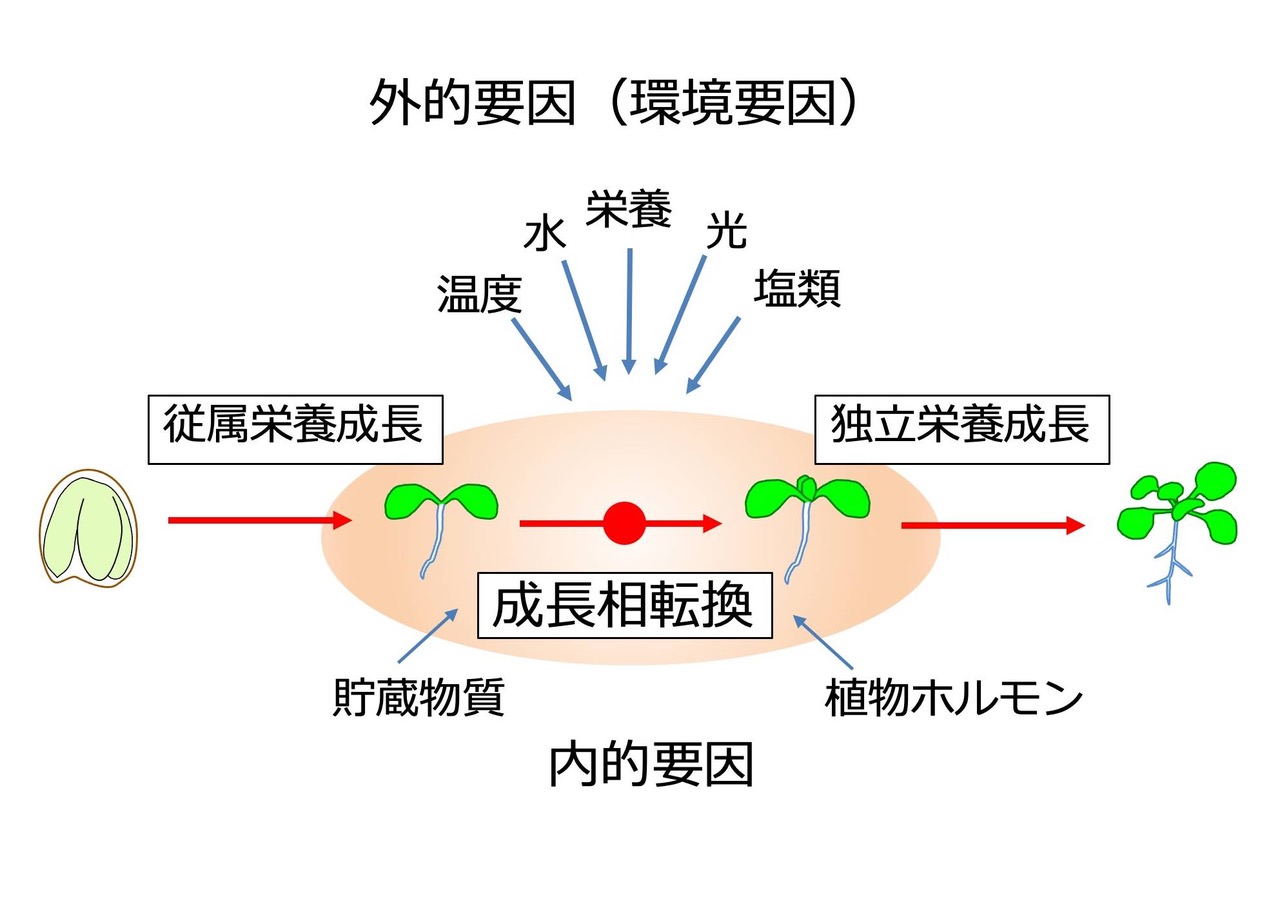
種子植物の芽生えは、発芽後に種子に蓄えられた栄養を使って成長し、光合成能力を獲得します。この過程は「実生の確立(seedling establishment)」と呼ばれ、従属栄養成長から独立栄養成長への成長相の転換です。この成長ステージの移行には、貯蔵物質や植物ホルモンなどの内的要因と、光や水分、ミネラルなどの外的要因(環境要因)が関わっています(図2)。
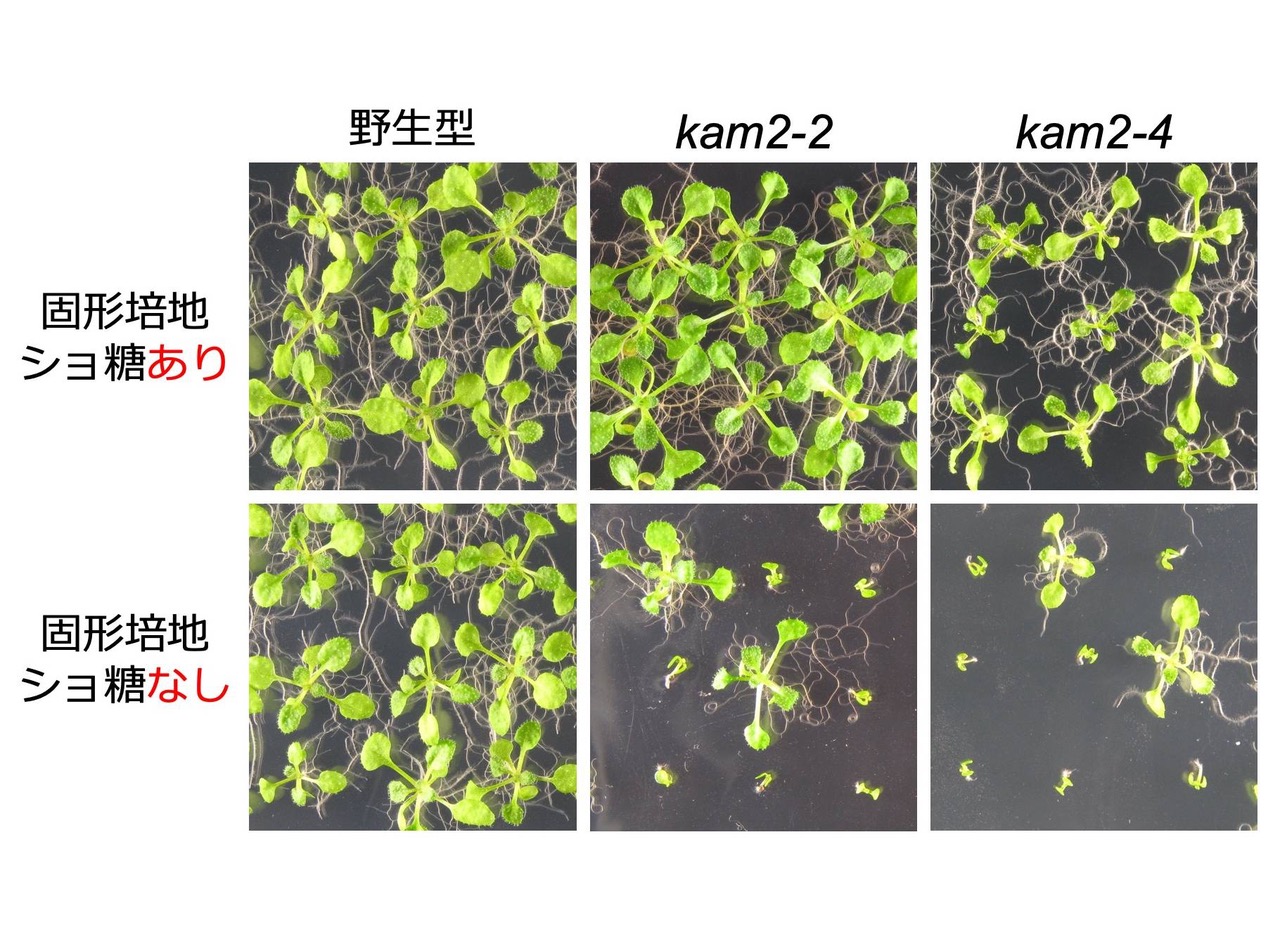
これまでの先行研究で、モデル植物シロイヌナズナ(Arabidopsis thaliana)の一部の変異体は、ショ糖を含まない固形培地で発芽させると、ある割合で実生の確立に失敗し、子葉(芽生えの初期の葉)を広げたステージで成長が止まってしまう芽生えが生じることが分かっていました(図3)。この芽生えの成長停止の原因は、脂質などの栄養不足や代謝の異常が考えられていましたが、詳しいことは分かっていませんでした。
最近の私たちの研究から、ショ糖を含まない固形培地での芽生えの成長停止は、固形培地のゲル濃度を高くしたり、培地の表面にプラスチックシートを敷くことで和らげられ、植物の地上部と培地との接触が引き金になっていることが明らかになってきました。さらに、この成長停止は、シロイヌナズナの一部の変異体だけでなく、野生型でも起こる可能性のある普遍的な環境応答であることが分かりました。
大地に根を張る動かない植物は、周囲の環境に適応する能力をもっています。この研究は、植物がどのように環境に適応しているのかについて新しい理解をもたらし、今後の研究で植物が環境に適応するメカニズムをさらに解き明かす手がかりになると期待されます。
最近の主な発表論文
- Takagi J, Takahashi H, Moriya KC, Nagano M, Fukao Y, Ueda H, Tamura K, Shimada T, Hara-Nishimura I. (2025) Plant-specific tail-anchored coiled-coil protein MAG3 stabilizes Golgi-associated ERESs to facilitate protein exit from the ER. Commun Biol. 8(1), 358, doi: 10.1038/s42003-025-07602-1.
- 守屋 健太、嶋田 知生. (2024) 陸上植物の組織形成を制御するbHIH型転写因子の機能進化. Plant Morphol. 36(1), 69-76.
- Chen Y, Nishimura K, Tokizawa M, Yamamoto YY, Oka Y, Matsushita T, Hanada K, Shirai K, Mano S, Shimizu T, and Masuda T. (2024) Cytosolic heme catabolism by alternative localization of heme oxygenase 1 in plant cells. Plant Physiol. 195 2937-2951, doi: 10.1093/plphys/kiae288.
- Seki M, Kuze Y, Zhang X, Kurotani KI, Notaguchi M, Nishio H, Kudoh H, Suzaki T, Yoshida S, Sugano S, Matsushita T, Suzuki Y. (2024) An improved method for the highly specific detection of transcription start sites. Nucleic Acids Res. 52(2):e7, doi: 10.1093/nar/gkad1116.
- Hosokawa C, Yagi H, Segami S, Nagano AJ, Koumoto Y, Tamura K, Oka Y, Matsushita T, Shimada T. (2024) The Arabidopsis katamari2 Mutant Exhibits a Hypersensitive Seedling Arrest Response at the Phase Transition from Heterotrophic to Autotrophic Growth. Plant Cell Physiol. 65(3) 350-361, doi: 10.1093/pcp/pcad156.
- Shimada T, Moriya KC. (2023) Co-option of stomatal bHLH genes drives development of the seta in Marchantia. Nature Plants. 9, 207-208.
- Kobayashi H, Murakami K, Sugano SS, Tamura K, Oka Y, Matsushita T, Shimada T. (2023) Comprehensive analysis of peptide-coding genes and initial characterization of an LRR-only microprotein in Marchantia polymorpha. Front Plant Sci. 13, 1051017.
- Moriya KC, Shirakawa M, Loue-Manifel J, Matsuda Y, Lu YT, Tamura K, Oka Y, Matsushita T, Hara-Nishimura I, Ingram G, Nishihama R, Goodrich J, Kohchi T, Shimada T. (2023) Stomatal regulators are co-opted for seta development in the astomatous liverwort Marchantia polymorpha. Nature Plants. 9, 302-314.
- Kozuka T, Oka Y, Kohzuma K, Kusaba M. (2023) Cryptochromes suppress leaf senescence in response to blue light in Arabidopsis. Plant Physiol. 191, 2506-2518.
- Takeda T, Shirai K, Kim YW, Higuchi-Takeuchi M, Shimizu M, Kondo T, Ushijima T, Matsushita T, Shinozaki K, Hanada K. (2023) A de novo gene originating from the mitochondria controls floral transition in Arabidopsis thaliana. Plant Mol Biol. 111, 189-203.
- Hu S, Li B, Wu F, Zhu D, Zouhar J, Gao C, Shimada T, Rojo E, Hara-Nishimura I, Jiang L, Shen J. (2022) SPlant ESCRT protein ALIX coordinates with retromer complex in regulating receptor-mediated sorting of soluble vacuolar proteins. Proc. Natl Acad Sci USA 119, e2200492119.
- Yagi H, Tamura K, Matsushita T, Shimada T. (2021) Spatiotemporal relationship between auxin dynamics and hydathode development in Arabidopsis leaf teeth. Plant Signal. Behav. 16, 1989216.
- Yagi H, Nagano AJ, Kim J, Tamura K, Mochizuki N, Nagatani A, Matsushita T, Shimada T. (2021) Fluorescent protein-based imaging and tissue-specific RNA-seq analysis of Arabidopsis hydathodes. J Exp. Bot. 72, 1260-1270.
- Sakoda K, Yamori W, Shimada T, Sugano SS, Hara-Nishimura I, Tanaka Y. (2021) Higher Stomatal Density Improves Photosynthetic Induction and Biomass Production in Arabidopsis Under Fluctuating Light. Front Plant Sci. 11, 589603.
- Nomoto M, Skelly MJ, Itaya T, Mori T, Suzuki T, Matsushita T, Tokizawa M, Kuwata K, Mori H, Yamamoto YY, Higashiyama T, Tsukagoshi H, Spoel SH, and Tada Y. (2021) Suppression of MYC transcription activators by the immune cofactor NPR1 fine-tunes plant immune responses. Cell Rep. 37, 110125.
- Izuishi Y, Isaka N, Li H, Nakanishi K, Kageyama J, Ishikawa K, Shimada T, Masuta C, Yoshikawa N, Kusano H, Yazaki K. (2020) Apple latent spherical virus (ALSV)-induced gene silencing in a medicinal plant, Lithospermum erythrorhizon. Scientific Rep. 10, 13555.
- Takagi T, Kimori Y, Shimada T, Hara-Nishimura I. (2020) Fluorescent protein-based imaging and tissue-specific RNA-seq analysis of Arabidopsis hydathodes. iScience 23, 101265.
- Ichino T, Maeda K, Hara-Nishimura I, Shimada T. (2020) Arabidopsis ECHIDNA protein is involved in seed coloration, protein trafficking to vacuoles, and vacuolar biogenesis. J Exp. Bot. 71, 3999-4009.
- Ishishita K, Higa T, Tanaka H, Inoue SI, Chung A, Ushijima T, Matsushita T, Kinoshita T, Nakai M, Wada M, Suetsugu N, and Gotoh E. (2020) Phototropin 2 contributes to the chloroplast avoidance response at the chloroplast-plasma membrane interface. Plant Physiol. 183, 304-316.
- Matsushita T. (2019) Regulation of alternative splicing by phytochromes. Methods Mol. Biol. 111, 189-203.
- Ishikawa K, Tamura K, Fukao Y, Shimada T. (2019) Structural and functional relationships between plasmodesmata and plant endoplasmic reticulum-plasma membrane contact sites consisting of three synaptotagmins. New Phytologist. 226, 798-808.
- Sakaguchi J, Matsushita T, and Watanabe Y. (2019) DWARF4 accumulation in root tips is enhanced via blue light perception by cryptochromes. Plant Cell Environ. 42, 1615-1629.
- Nakazaki A, Yamada K, Kunieda T, Tamura K, Hara-Nishimura I, Shimada T. (2019) Biogenesis of leaf endoplasmic reticulum body is regulated by both jasmonate-dependent and independent pathways. Plant Signal. Behav. 14, 1622982.
- Shimada TL, Shimada T, Okazaki Y, Higashi Y, Saito K, Kuwata K, Oyama K, Kato M, Ueda H, Nakano A, Ueda T, Takano Y, Hara-Nishimura I. (2019) HIGH STEROL ESTER 1 is a key factor in plant sterol homeostasis. Nature Plants 5, 1154-1166.
- Maeda K, Kunieda T, Tamura K, Hatano K, Hara-Nishimura I, Shimada T. (2019) Identification of periplasmic root-cap mucilage in developing columella cells of Arabidopsis thaliana. Plant Cell Physiol. 60, 1296-1303.
- Yoshinari A, Hosokawa T, Amano T, Beier MP, Kunieda T, Shimada T, Hara-Nishimura I, Naito S, Takano J. (2019) Polar Localization of the Borate Exporter BOR1 Requires AP2-Dependent Endocytosis. Plant Physiol. 179, 1569-1580
- Nakazaki A, Yamada K, Kunieda T, Sugiyama R, Hirai MY, Tamura K, Hara-Nishimura I, Shimada T. (2019) Leaf endoplasmic reticulum bodies identified in Arabidopsis rosette leaves are involved in defense against herbivory. Plant Physiol. 179, 1515-1524.
- Ishikawa K, Tamura K, Shimada T. (2018) Subcellular localisation of an endoplasmic reticulum-plasma membrane tethering factor, SYNAPTOTAGMIN 1, is affected by fluorescent protein fusion. Plant Signal. Behav. 13, e1547577.
- Sugano SS, Nishihama R, Shirakawa M, Takagi J, Matsuda Y, Ishida S, Shimada T, Hara-Nishimura I, Osakabe K, Kohchi T. (2018) Efficient CRISPR/Cas9-based genome editing and its application to conditional genetic analysis in Marchantia polymorpha. PLoS One 13, e0205117.
- Ishikawa K, Tamura K, Ueda H, Ito Y, Nakano A, Hara-Nishimura I, Shimada T. (2018) Synaptotagmin-Associated Endoplasmic Reticulum-Plasma Membrane Contact Sites Are Localized to Immobile ER Tubules. Plant Physiol. 178, 641-653.
- Shimada T, Kunieda T, Sumi S, Koumoto Y, Tamura K, Hatano K, Ueda H, Hara-Nishimura I. (2018) The AP-1 Complex is Required for Proper Mucilage Formation in Arabidopsis Seeds. Plant Cell Physiol. 59, 2331-2338.
- Shimada T, Fuji K, Ichino T, Teh OK, Koumoto Y, Hara-Nishimura I. (2018) GREEN FLUORESCENT SEED, to Evaluate Vacuolar Trafficking in Arabidopsis Seeds. Methods Mol Biol., 1789, 1-7.
- Shimada T, Takagi J, Ichino T, Shirakawa M, Hara-Nishimura I. (2018) Plant Vacuoles. Annu Rev Plant Biol. 69, 123-145.
- Ushijima T, Hanada K, Gotoh E, Yamori W, Kodama Y, Tanaka H, Kusano M, Fukushima A, Tokizawa M, Yamamoto YY, Tada Y, Suzuki Y, Matsushita T. (2017) Light controls protein localization through phytochrome-mediated alternative promoter selection. Cell 171, 1316-1325.
- Sakai Y, Sugano SS, Kawase T, Shirakawa M, Imai Y, Kawamoto Y, Sugiyama H, Nakagawa T, Hara-Nishimura I, Shimada T. (2017) The chemical compound bubblin induces stomatal mispatterning in Arabidopsis by disrupting the intrinsic polarity of stomatal lineage cells. Development 144, 499-506.
- Wang Q, Zuo Z, Wang X, Gu L, Yoshizumi T, Yang Z, Yang L, Liu Q, Liu W, Han Y, Kim J, Liu B, Wohlschlegel JA, Matsui M, Oka Y, Lin C. (2016) Photoactivation and inactivation of Arabidopsis cryptochrome 2. Science 354, 343-347.
- Shikata H, Hanada K, Ushijima T, Nakashima M, Suzuki Y, Matsushita T. (2014) Phytochrome controls alternative splicing to mediate light responses in Arabidopsis. Proc Natl Acad Sci USA. 111, 18781-18786.
- Sugano SS, Shimada T, Imai Y, Okawa K, Tamai A, Mori M, Hara-Nishimura I. (2010) Stomagen positively regulates stomatal density in Arabidopsis. Nature 463, 241-244.
最近の受賞
2024 年度学位論文
修士論文
- 島 考元「シロイヌナズナにおける順遺伝学を起点としたECT11の分子機能解析」
メンバー
- 松下 智直(教授)
- 嶋田 知生(講師)
- 岡 義人(助教)
- 神山 佳明(日本学術振興会特別研究員PD)
- 井上 亜美(技術補佐員)
- 三嶋 文(技術補佐員)
- 河本 恭子(教務補佐員)
- 三星 亮太朗(博士後期課程3年)
- 村上 吉朗(博士後期課程2年)
- 古賀 大翔(修士課程2生年)
- 岩島 亜季(修士課程2年)
- 元谷 涼太郎(学部4回生)